Shock AI Discovery sugiere que todavía no hemos descubierto la mitad de lo que hay dentro de nuestras células.
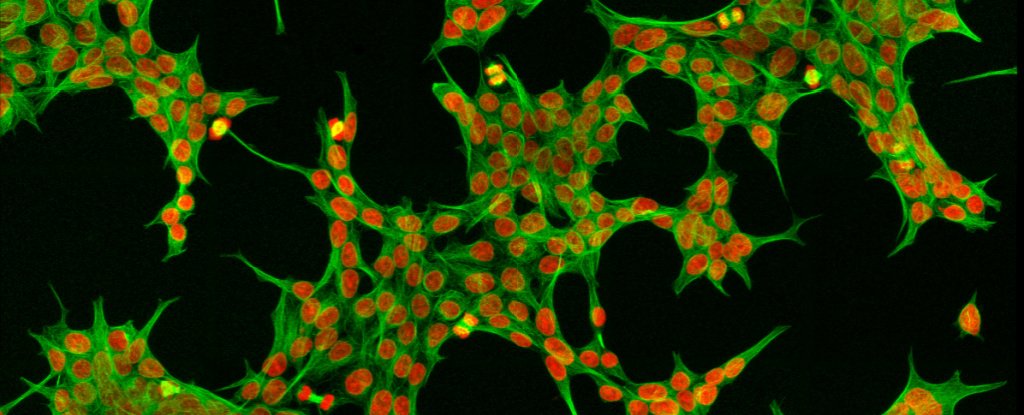
Dentro de cada célula del cuerpo humano hay una constelación de proteínas, millones de ellos. Todos están empujándose, siendo rápidamente ensamblados, doblados, empaquetados, enviados, cortados y reciclados en un hervidero de actividad que corre a un ritmo febril para mantenernos vivos y funcionando.
Pero sin un inventario completo del universo de proteínas dentro de nuestras células, los científicos se ven presionados a evaluar a nivel molecular qué es lo que está mal en nuestro cuerpo y que conduce a la enfermedad.
Ahora, los investigadores han desarrollado una nueva técnica que utiliza inteligencia artificial asimilar datos de imágenes microscópicas de células individuales y análisis bioquímicos, para crear un «mapa unificado» de componentes subcelulares, la mitad de los cuales, al parecer, nunca antes habíamos visto.
«Los científicos se dieron cuenta hace mucho tiempo de que hay más cosas que no sabemos de las que sabemos, pero ahora finalmente tenemos una manera de mirar más a fondo». dice científico informático y biólogo de redes Trey Ideker, Universidad de California (UC) San Diego.
Los microscopios, por muy poderosos que sean, permiten a los científicos observar el interior de las células individuales, hasta el nivel de orgánulos como las mitocondrias, los paquetes de energía de las células, y los ribosomas, las fábricas de proteínas. Incluso podemos agregar tintes fluorescentes para marcar y rastrear proteínas fácilmente.
Las técnicas bioquímicas pueden profundizar aún más, perfeccionando las proteínas individuales utilizando, por ejemplo, anticuerpos que se unen a la proteína, la extraen de la célula y observan qué más se le atribuye.
La integración de estos dos enfoques es un desafío para los biólogos celulares.
«¿Cómo se llena este vacío de la escala nanométrica a la micra? Este ha sido un gran obstáculo en las ciencias biológicas», agregó. explicar Ideker
«Resulta que puedes hacer eso con inteligencia artificial: ver datos de múltiples fuentes y pedirle al sistema que los monte en un modelo celular».
El resultado: Ideker y sus colegas invierten mapas didácticos de células globulares que nos dan una vista de pájaro de orgánulos de colores en una intrincada red de interacciones proteína-proteína, organizadas por pequeñas distancias entre ellos.

Combinando datos de imágenes de una biblioteca llamada Human Protein Atlas y mapas existentes de interacciones de proteínas, el aprendizaje automático El algoritmo se encargó de calcular las distancias entre pares de proteínas.
El objetivo era identificar comunidades de proteínas, llamadas ensamblajes, que coexisten en células a diferentes escalas, desde las muy pequeñas (menos de 50 nm) hasta las muy ‘grandes’ (más de 1 μm).
El algoritmo clasificó a una tímida de las 70 comunidades de proteínas, que se entrenó utilizando una biblioteca de referencia de proteínas con diámetros conocidos o estimados y se validó con experimentos adicionales.
Aproximadamente la mitad de los componentes proteicos identificados aparentemente son desconocidos para la ciencia, nunca documentados en la literatura publicada, los investigadores sugerir.
En la mezcla había un grupo de proteínas que formaban una estructura desconocida, que los investigadores encontraron probablemente era responsable de la enmienda y cortar transcripciones frescas del código genético que se utilizan para producir proteínas.
Otras proteínas mapeadas incluyeron sistemas de transporte transmembrana que bombean suministros dentro y fuera de las células, familias de proteínas que ayudan a organizar cromosomas voluminosos y complejos de proteínas cuyo trabajo es producir, digamos, más proteínas.
Sin embargo, un gran esfuerzo no es la primera vez que los científicos intentan mapear el funcionamiento interno de las células humanas.
Otros esfuerzos para crear mapas de referencia de interacciones de proteínas han dado como resultado números igualmente alucinantes y probé medir los niveles de proteína a través de los tejidos del cuerpo humano.
Los investigadores también han desarrollado técnicas para visualizar y rastrear la interacción y el movimiento de las proteínas en las células.
Este estudio piloto va un paso más allá, aplicando el aprendizaje automático a imágenes de microscopía celular que ubican proteínas en relación con puntos de referencia de células grandes, como el núcleo, y datos de estudios de interacción de proteínas que identifican los vecinos más cercanos de una proteína en la nanoescala.
«La combinación de estas tecnologías es única y poderosa porque es la primera vez que se combinan mediciones a escalas muy diferentes». dice Yue Qin, hombre de bioinformática, también de UC San Diego.
Al hacerlo, la técnica de células integradas multiescala o MuSIC «aumenta la resolución de la imagen al tiempo que otorga una dimensión espacial a las interacciones de proteínas, allanando el camino para incorporar diversos tipos de datos en mapas de células en todo el proteoma», Qin, Ideker y colegas. escribir.
Para ser claros, esta investigación es muy preliminar: el equipo se centró en validar su método y solo analizó los datos disponibles sobre 661 proteínas en un tipo de célula, una línea de células renales que los científicos han estado cultivando en el laboratorio durante cinco décadas.
Los investigadores planean aplicar su técnica innovadora a otros tipos de células, dice Ideker
Pero mientras tanto, tendremos que aceptar humildemente que somos meros intrusos dentro de nuestras propias células, capaces de comprender una pequeña fracción del total. proteoma.
«Con el tiempo, es posible que podamos comprender mejor la base molecular de muchas enfermedades, comparando las diferencias entre las células sanas y las enfermas». dice Ideker
El estudio fue publicado en Naturaleza.





