El equipo desarrolla el primer sistema libre de células en el que la información genética y el metabolismo trabajan juntos
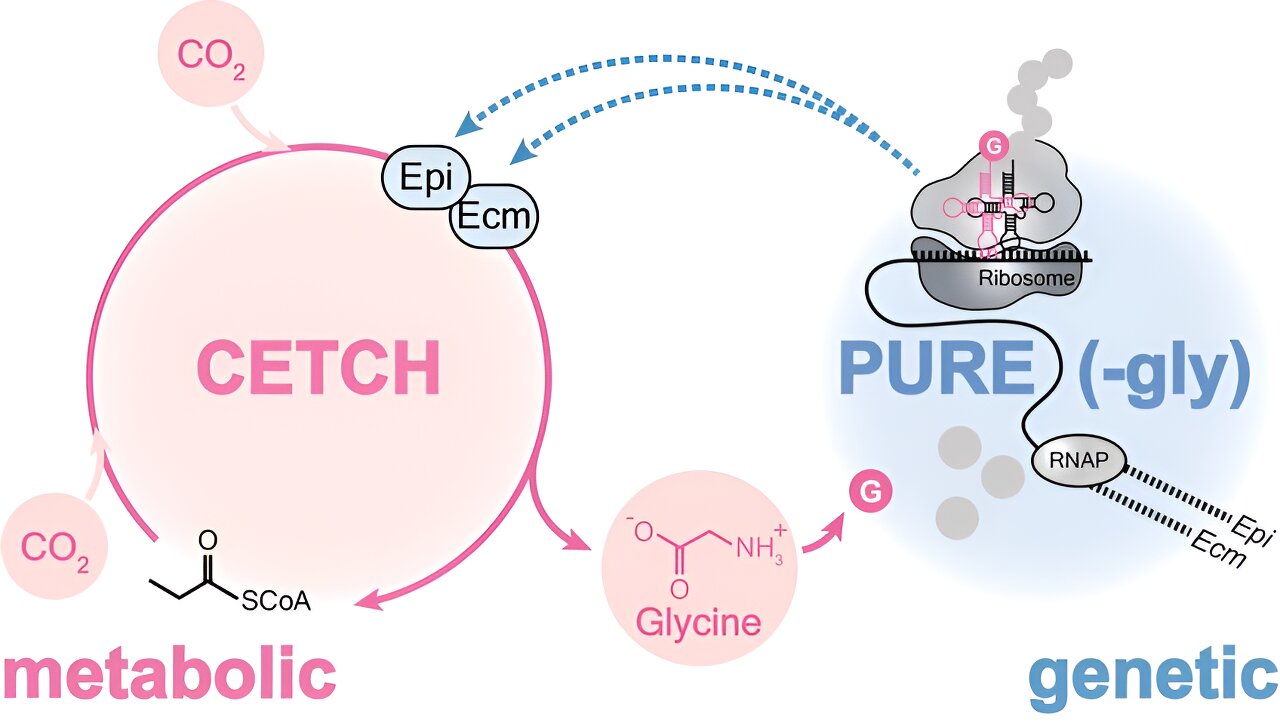
Sistema libre de células compuesto por niveles metabólicos (ciclo Cetch, rosa) y genéticos (puro, azul) interdependientes que interactúan recursivamente entre sí. Pure produce las enzimas que faltan para el COdos fijación (es decir, Epi y Ecm) mediante transcripción y traducción (TX-TL) de los genes Epi y Ecm; Cetch utiliza estas enzimas para sintetizar glicina a partir de COdosapoyando así la producción de proteínas. Las abreviaturas de enzimas Epi, Ecm y RNAP significan metilmalonil-/etilmalonil-CoA epimerasa, etilmalonil-CoA mutasa y ARN polimerasa, respectivamente. Crédito: MPI f. Microbiología Terrestre/ Giaveri
La capacidad de todos los sistemas vivos para desarrollarse, organizarse y sostenerse se basa en un proceso cíclico en el que los genes y el metabolismo interactúan en paralelo. Mientras que los genes codifican los componentes del metabolismo, el metabolismo proporciona la energía y los componentes básicos para mantener y procesar la información genética.
En biología sintética, los investigadores exploran los principios de la vida reconstruyendo sus sistemas desde abajo hacia arriba, comenzando con el número mínimo de piezas necesarias. En los últimos años, este enfoque ha hecho posible desarrollar redes metabólicas complejas y sistemas genéticos libres de células fuera del entorno celular (in vitro), por ejemplo, en cámaras de microfluidos.
Lo que estos enfoques tienen en común es que todos los biocatalizadores que funcionan en estos sistemas se añaden desde el exterior, y todo el proceso sólo continúa mientras se le suministre un flujo continuo de nuevos componentes básicos, información y energía.
Al unir los niveles metabólico y genético, los investigadores quieren crear sistemas biológicos sintéticos autosostenibles que puedan generar sus propios componentes básicos e impulsar procesos recíprocamente, como en las células vivas. Un equipo dirigido por Tobias Erb del Instituto Max Planck de Microbiología Terrestre en Marburg, Alemania, ha dado un paso significativo hacia este objetivo.
El equipo desarrolló el primer sistema libre de células en el que una red genética y metabólica se mantienen funcionando mutuamente. El sistema produce enzimas metabólicas por sí solo y funciona tanto en el tubo de ensayo como en imitaciones celulares artificiales. Se basa en el ciclo sintético Cetch, una red metabólica que utiliza COdos como materia prima para producir moléculas orgánicas.
el trabajo es Publicado en el diario Ciencia.
El truco: interdependencia
«Acoplamos el ciclo Cetch con un sistema genético existente llamado Pure, una máquina de transcripción y traducción sintética que funciona con una mezcla de ribosomas, ADN, ARN y proteínas, fuera de las células vivas. Diseñamos los dos niveles para que funcionen juntos como un solo motor. Una vez iniciado, continúa porque las dos redes se retroalimentan», explica Simone Giaveri, becaria de EMBO y primera autora del artículo.
Para que esto funcione, los investigadores hicieron que los componentes dependieran unos de otros. Programaron Pure para producir dos de las enzimas Cetch. Sin embargo, esta variante pura carece del aminoácido esencial glicina, que es necesario para producir proteínas. Cetch ha sido modificado para producir glicina directamente a partir de COdos. Dado que Pure obtiene glicina de Cetch, el ciclo está cerrado.
Para demostrar que su enfoque funcionó, los investigadores primero agregaron glicina a Pure, que contenía la información para producir una proteína fluorescente. Su brillo indicaba la buscada actividad de la red genética. El siguiente paso fue introducir el ciclo Cetch sintético. Una vez que se introdujo la vía sintética, el sistema acoplado se volvió capaz de producir la propia glicina y, a su vez, dos proteínas Cetch, además de la proteína fluorescente.
De las más de 50 proteínas del sistema, el sistema sólo produce dos por sí solo. Sin embargo, esto es todo lo que se necesita para impulsar el ciclo sintético.
«Sin el componente genético y la retroalimentación mutua, el ciclo sólo duraría menos de una hora. El hecho de que sea autorregenerante significa que durará al menos doce horas antes de que el sistema se detenga por diversas razones, por ejemplo porque fallan los componentes o los subproductos se acumulan demasiado», explica Giaveri. «Hay que empezar con una cantidad mínima de glicina y así se continuará».
La mayoría de los elementos del metabolismo sintético todavía proceden del exterior.
«Aún estamos muy lejos de un sistema que pueda regenerar todos sus propios componentes», afirma Erb. Esto implicaría codificar redes metabólicas completas, codificar programas de autorreparación para extender la vida útil de los sistemas in vitro, así como integrar ciclos de reciclaje bioquímico.
“Hasta ahora sólo hemos podido producir un componente básico y todavía estamos muy lejos de poder producir todos los componentes básicos a partir de COdos. Sin embargo, hemos desarrollado un sistema operativo básico que se beneficiará de futuros desarrollos en este campo de investigación en rápido movimiento. Si miramos aún más hacia el futuro, podemos imaginar que en el futuro podremos hacer funcionar un sistema de este tipo con electricidad ligera o incluso sostenible”.
Un sistema operativo básico para futuros sistemas sostenibles
La orquestación de más de 50 proteínas, fuentes de energía, información genética y componentes básicos es el resultado de una enorme cantidad de experimentos en los que Giaveri probó y optimizó combinaciones en paralelo. Cada elemento del complejo sistema de Giaveri está diseñado con precisión para su propósito.
«Nuestro sistema se puede utilizar como unidad operativa, como motor básico para sistemas in vitro», afirma Erb. «Y como se basa en COdosEsto sería posible de forma totalmente sostenible, ya que esta materia prima está disponible en cantidades prácticamente ilimitadas».
Mas informaciones:
Simone Giaveri et al, Traducción y metabolismo integrados en una red bioquímica parcialmente autosintética, Ciencia (2024). DOI: 10.1126/ciencia.adn3856
Cita: El equipo desarrolla el primer sistema libre de células en el que la información genética y el metabolismo trabajan juntos (2024, 11 de julio) recuperado el 11 de julio de 2024 de https://phys.org/news/2024-07-team-cell -free-genetic- metabolismo.html
Este documento está sujeto a derechos de autor. Aparte de cualquier uso legítimo para estudios privados o fines de investigación, ninguna parte puede reproducirse sin permiso por escrito. El contenido se proporciona únicamente con fines informativos.




